Introduction
La portée de ce rapport est d'exposer les premiers pas de Web Scrapping avec R.
Qu'est-ce que c'est?
Le Web scrapping est une technique d'extraction du contenu de sites Web. Des applications sembent régulièrement proposer des solutions. Cependant, il est important de ne pas oublier que produire des données, peut s'avérer long couteux ou fastidieux. Ces pratiques sont souvent assimilées ? des m?thodes de pillage, ce gerne d'application ne vie jamais longtemps.
Les progr?és réalisés en matière d'ouverture des données et de politique d'Open Data promettent à l'étudiant en statistiques un nouveau champs d'analyse. Aurevoir les données Iris et Ozonne de R, les API donnent maintenant l'air d'offir un nouveau champ du possible avec des données autrefois jalousement gardées.
Toutefois, il s'avère que ces données ne sont pas si facilement récupérable. Rencontrées lors de notre exploration sur l'API Bordeaux Métropole ces données sont stocké sous des formats farfellus ne permetant ni lecture, ni récupération massive. Des exemples de script parcourant la source de ces données nous indiquent cependant qu'il est possible de les récupérer. Challenge Accepted.
Packages
Le voici, le package de Web scrapping sous R dont nous nous sommes servi:
library(rvest)
Inspir? de librairies pour python, il permet de racler les informations des pages web.
library(xml2)
library(XML)
Ils permetent de traiter des objets HTML/XML, indispensable puisque les donn?es sur les site web seront en XLM brut.
library(stringr)
library(dplyr)
stringr est un package trés efficace lorsqu'on cherche à manipuler des chaînes de caractère, dplyr est pratique pour la manipulation des dataframes. Et readr promet de faire gagner de précieuses secondes dans l'?criture et la lecture des données.
Initialisation
Pour appuyer notre explication, nous avons choisi le site theses.fr. Vous devez repérer sur le site de votre choix le "search query". Par exemple sur google (ne le faites pas avec google, ils ne seraient pas content), le search query est la requ?te que vous lui demander: c'est le QUERY dans: https://google.fr/search?q=[QUERY]. Pour theses.fr, par exemple en souhaitant parcourir tout les idividus de la base:
q <- str_c("http://www.theses.fr/personnes/?q=","fq=dateSoutenance:[1984-01-01T23:59:59Z%2BTO%2B2016-12-31T23:59:59Z]", # possibilité de set query
"start=0&status=&access=&prevision=",
"filtrepersonne=",
"zone1=titreRAs",
"val1=&op1=AND",
"zone2=auteurs",
"val2=&op2=AND",
"zone3=etabSoutenances",
"val3=&op3=AND",
"zone4=dateSoutenance",
"val4a=",
"val4b=",
"type=",
"format=xml", # pas pr?sent dans notre query
sep = "&"
)
Le format XML à la fin du query est à rajouter dans l'URL et à vérifier si ca fonctionne. La littérature prétends que aujourd'hui l' HTML est une norme mourante car destin? presque exclusivement au web. XML hierarchise les données, on le vera plus loin, trés utile à l'echange de celle-ci. En principe, les développeurs web utilisent maintenant le XHTML qui est à mis chemin, et qui est plus conforme aux normes XML. En théorie vous devriez donc trouver facilement des pages de métadata en HTML qui s'exportent en XML.
Deux possibilités s'offrent à vous. Soit vous avez peu de données (= peu de pages), vous pouvez alors faire tourner le script à la main. Soit, comme nous vous avez un nombre déraisonnable de pages, pas d'autres choix de faire boucler le script. Des bases en XML sont nécessaires. Le plus simple lorsqu'on a en encore jamais rencontré ce language, est d'ouvrir une page en XML et de regarder comment elle est construite. Les informations sont stocké sous leurs formes d'origines entre des balises (nodes). L'accés à celle ci ce fera par l'appel de ces balises.
<result name="response" numFound="14" start="0">
<doc>
<str name="personne">Jeremie Bigot</str>
<str name="personneNP">Bigot Jeremie</str>
<date name="dateInsert">27-08-2015</date>
<date name="dateMaj">05-09-2016</date>
<str name="ppn">null</str>
<str name="actif">oui</str>
<arr name="thesesEnTantQueDirecteur">
<str name="nbTheses">1</str>
<arr name="numThesesEnTantQueDirecteur">
<str>s138623</str>
</arr>
<arr name="titreThesesEnTantQueDirecteur">
<str>Propriétés statistiques du barycentre dans l'espace de Wasserstein et algorythmes rapides pour le transport optimal</str>
</arr>
</arr>
</doc>
Commencons par créer un vecteur parcourant les pages. Dans notre cas, on a 10 résultats par page, on recoupe donc le nombre totale de résultat par intervalles de 10. On accéde au balise plutôt facilement grâce au fonction de la library rvest. Nos conclusions sont que balise est la traduction choisie pour nodes. Ainsi, dans notre cas, on récupére aisement le nombre total de résultats car c'est quelque chose qui est inscrit lorsqu'on fait une requète sur le site. C'est souvent le cas à priori, google donne même le temps qu'il lui a fallu pour trouver tout ces résultats.
j <- read_xml(q) %>%
xml_node("result") %>%
xml_attr("numFound") %>%
as.integer %>%
seq(0, ., by = 10)
Loop
Afin de s'assurer qu'on parcourt toutes les pages, on choisit d'enlever les pages qu'on a déjà parcouru au fur et à mesure, et de commencer notre script par un while(length(j)>0). Ensuite, on applique un sample à j, afin de prendre les résultats dans un ordre aléatoire et ne pas favoriser un rangement des resultats par un certain ordre. Comme il n'est pas trés pertinent de récupérer les 616 660 résultats, pour des raisons de stockage et manipulation. Nous écrivons le csv à chaque itération, pour assurer la création de celui-ci. Voici donc par exemple le code qui nous permet de récupérer la page, le nom de la personne, son identifiant ppn et si il est toujours actif.
p <- data_frame(
page = i %>% as.integer,
personne = xml_nodes(p, "str[name='personne']") %>% xml_text,
ppn = xml_nodes(p, "str[name='ppn']") %>% xml_text,
actif = xml_nodes(p, "str[name='actif']") %>% xml_text)
Problèmes eventuels
Cette dernière partie à pour objet de solutionner les problèmes que nous avons rencontrés au cours de nos implémentations.
Nos scripts se sont avérés trés long ( 35h pour 17 000 lignes) à tourner sur nos ordinateurs. Nous nous sommes alors intéressées à des alternatives. Bien que beaucoup plus rapide sur les ordinateurs du CREMI (service informatique), ce n'etait pas suffisant. La solution trouvée, était de venir faire tourner le code sur le serveur de la fac. En effet, si les ordinateurs sont éteins la nuit, le serveur n'est jamais étein. Pour envoyer un script tourner sur un serveur distant, nous avons trouvez une page qui l'expliquait plutôt bien. Il n'est juste pas préciser comment faire avec R: il faut enregistrer son code en .cpp (possible avec librarie Rcpp.h de C++ qui permette programmer en r), puis l'appeler sur le terminal ( une fois qu'il est passé en screen). On ne l'expliquera pas mieux que la documentation présente sur le net, nous vous conseillons plutôt de cherchez vers là.
Un autre soucis venant d'éventuelles valeurs manquante entre les balises.
Error in doc_parse_raw(x, encoding = encoding, base_url = base_url, as_html = as_html, :
xmlParseEntityRef: no name [68]
Pour la solutionner, il faut tester à chaque itération si la la lecture du xml a bien fonctionnée: notre boucle au final:
for (i in sample(j)) {
cat("[", "ESSAI", str_pad(i, 6, side = "left"), "]") # permet d'avoir de l'info sur ou en est la boucle
p <- try( # try() est dans la library de base de R
str_c("start=", i) %>% # permet de tester une requéte sans tout intérompre
str_replace(q, "start=0", .) %>%
read_xml,
silent = TRUE)
if (!"try-error" %in% class(p)) { # si la lecture est bonne
p <- data_frame(
page = i %>% as.integer,
personne = xml_nodes(p, "str[name='personne']") %>% xml_text,
ppn = xml_nodes(p, "str[name='ppn']") %>% xml_text,
actif = xml_nodes(p, "str[name='actif']") %>% xml_text#,
)
d <- rbind(d, p)
cat(":", nrow(d), "rows", sum(!is.na(d$ppn)), "PPNs\n")
write_csv(d, f)
} else {
cat(":", p[1])
}
}
Analyse
Une fois toutes ces données collectées nous avons fais un peu d'analyse.
Commençons par regarder la longueur des titres de thèses par discipline:
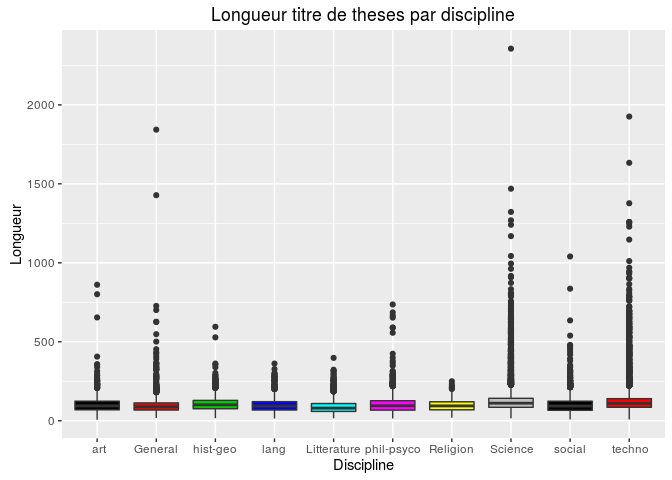
Sans surprise c'est les titres en science et technologie qui sont les
plus long. Maintenant si on s'intéresse de plus prés aux spécialités des
sciences:
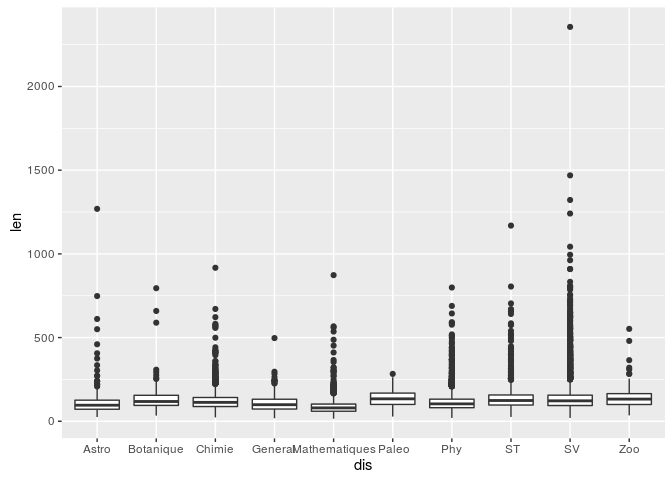
Il est aussi intéressant de voir l'évolution du nombres de théses par
discipline:
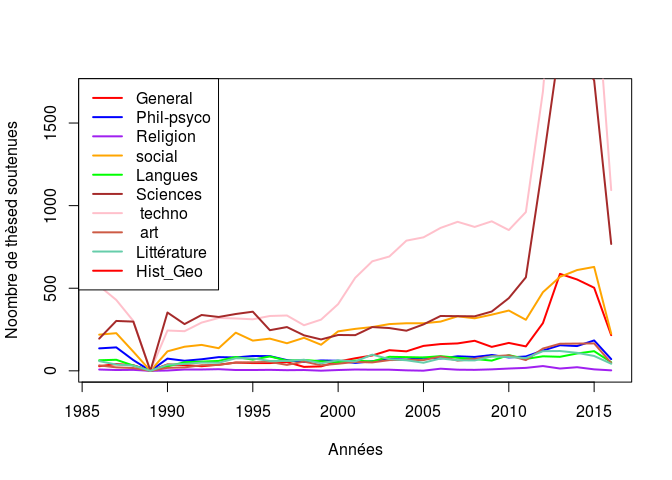
On remarque une explosion des siences et technologies ces dernières
années.
Et enfin, voici un network des disciplines:

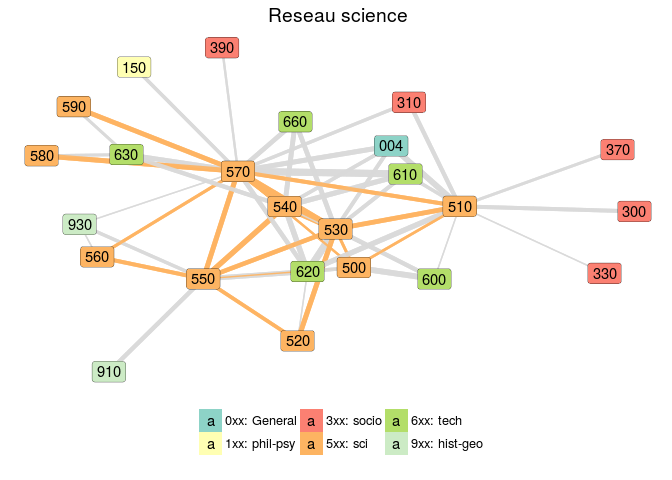
Documentation
Explication Web scraping avec python